中科院生态中心杨敏团队WR:污水病毒基因组监测揭示群体免疫驱动SARS-CoV-2加速演化
图文摘要
成果简介
近日,中国科学院生态环境研究中心杨敏研究组与中国疾病预防控制中心环境与健康相关产品安全所、清华大学环境学院合作,在Water Research上发表了题为“Population immunity enhances the evolution of SARS-CoV-2 in Beijing revealed by wastewater genomic surveillance”的研究论文(DOI: 10.1016/j.watres.2025.123649)。通过大规模污水采样与病毒基因组测序,构建了一个高分辨率的病毒演化监测体系,揭示了群体免疫如何影响区域SARS-CoV-2演化。与传统临床监测相比,污水病毒基因组监测在识别病毒变异株与基因突变方面显示出更高的灵敏度与覆盖度。在“动态清零”政策调整后,北京经历了第一波新冠人口集中感染,形成了强效的群体免疫屏障,有效阻断了国际关注的BQ.1和CH.1.1变异株的传播。该过程对区域病毒种群施加了显著的免疫选择压力,使北京的毒株进化速度较国际水平加快了12.5%。随着群体免疫水平的逐步衰退,XBB变异株在第二波疫情中取代BF.7.14成为优势毒株,随后EG.5在第三波中流行,毒株向抗体中和逃逸能力增加的方向快速进化。整体而言,北京高强度的群体感染加速了SARS-CoV-2的区域适应性进化,并通过群体免疫衰减与病毒进化的动态反馈机制,驱动优势毒株的快速更替。
引言
SARS-CoV-2自疫情暴发以来持续进化,多个变异株相继成为主导。病毒演化受突变本底、人群免疫与防控政策等多重因素影响。2022年底中国“动态清零”政策调整后,北京暴发了第一波大规模疫情,为病毒提供了快速进化的窗口。然而目前尚缺乏系统性的研究评估大规模集中感染事件对区域病毒进化的影响。污水病毒基因组监测作为反映区域病毒组成和动态变化的有效工具,具备高覆盖的优势。本研究长周期、多点位采集北京市政污水样本,开展病毒全基因组测序,系统分析了疫情防控政策调整后SARS-CoV-2的进化速率、变异特征及其与人群免疫水平之间的关系,为未来疫情预警与疫苗开发提供数据支持。
图文导读
毒株演替
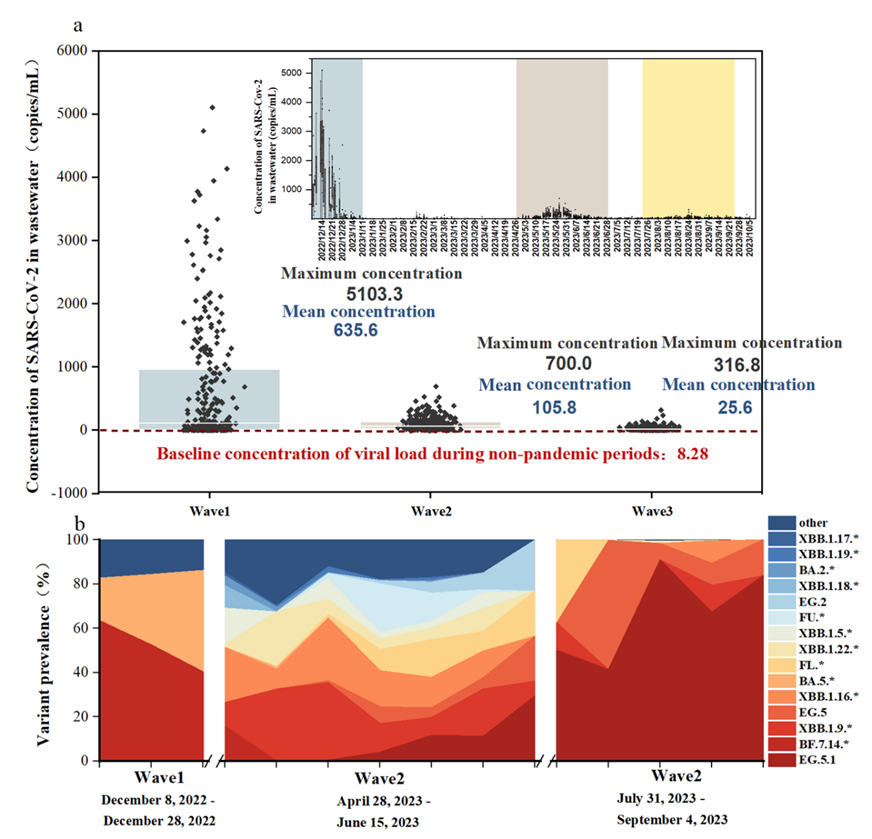
Fig. 1. Changes in abundance (a) and variant composition (b) of SARS-CoV-2 in wastewater of Beijing since December 2022. Copyright 2025, Elsevier Inc.
2022年12月至2023年10月,北京市污水中SARS-CoV-2浓度呈现出三次疫情波峰,对应三波流行浪潮。病毒浓度峰值从第一波的5103.3 copies/mL降至第三波的316.8 copies/mL,表明疫情强度逐步下降。与此同时,主导变异株从BF.7.14.*迅速演替为XBB系列,再到EG.5.1。
污水与临床监测的对比
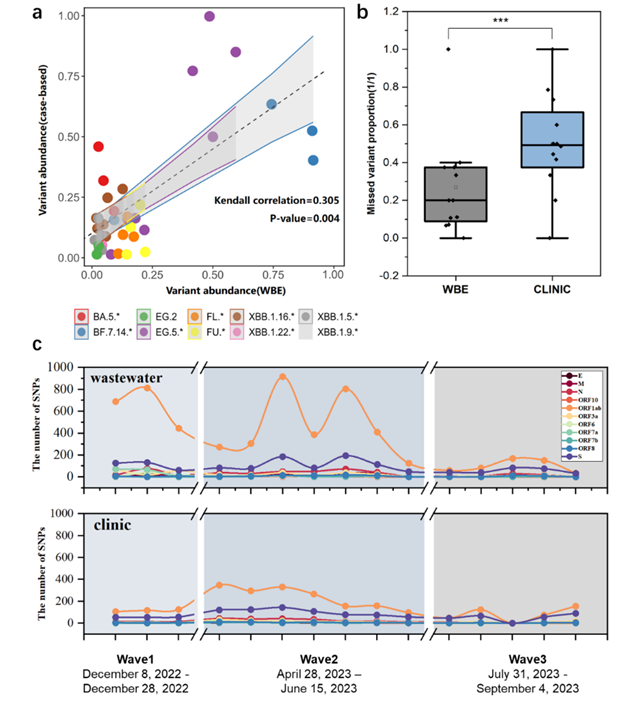
Fig. 2.Comparison of wastewater genome sequencing data with the clinical one. a. Correlation of the abundance of variants detected in wastewater with those detected in clinical cases. Variants with an abundance less than < 0.01 were excluded prior to the analysis; b. Proportion of missed variants in wastewater monitoring and clinical monitoring, with t-tests used for significance analysis, *** represents P < 0.001; c. Comparison of the SNPs’ abundance detected in wastewater variants (top) with that in clinical ones (bottom).Copyright 2025, Elsevier Inc.
对比同时期的1111例临床测序数据,污水监测共检测到244种变异株,是临床监测结果的2.46倍。污水数据中单核苷酸多态(SNP)数量在三波疫情中分别为临床数据的7.14、3.12与1.72倍,进一步证明了污水病毒基因组监测的高覆盖度。此外,两种监测方法得到的主要变异株相对丰度显著正相关(Kendall τ = 0.305, p < 0.01),验证了污水病毒监测与临床监测的高一致性,证明了污水病毒监测的结果可靠性。
区域与全球系统发育路径比较
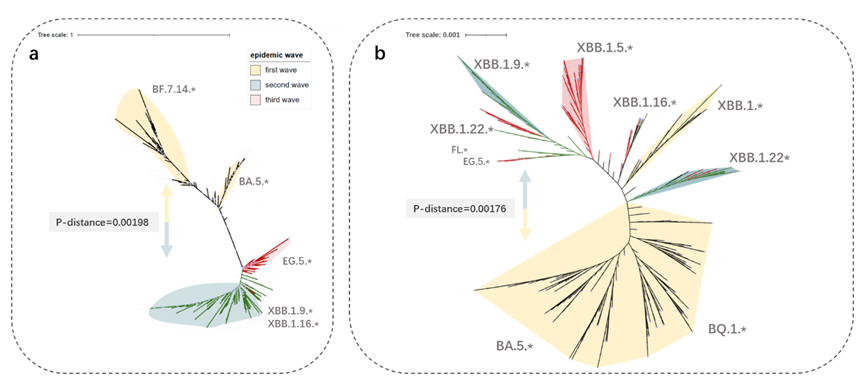
Fig. 3.The maximum likelihood phylogenetic trees constructed based on sequences and high-quality SARS-Cov-2 SNPs obtained from wastewater monitoring in (a) Beijing and (b) regions outside the Chinese mainland.Copyright 2025, Elsevier Inc.
系统发育分析显示,北京在第一波至第二波之间的毒株无明显重叠,BF.7.14.毒株直接被XBB系列取代,未经历国际流行毒株BQ.1与BN.*,而同时期的全球病毒演替呈连续进化的模式。北京病毒株在第一、二波之间的遗传距离显著高于同期国际水平,显示区域病毒进化速率快了12.5%。
正选择作用驱动关键蛋白进化
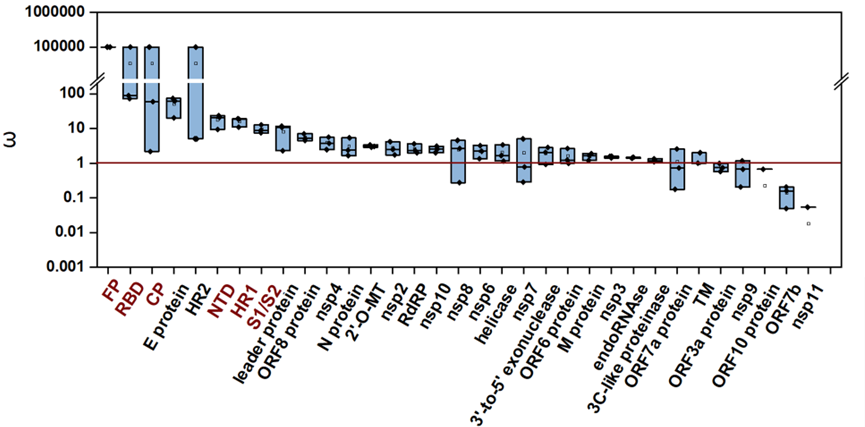
Fig. 4. The average ω values of different protein domains of the detected SARS-CoV-2 strains in wastewater samples. 2'-O-Ribose Methyltransferase: 2'-O-MT; RNA-dependent RNA Polymerase: RdRP; Nucleocapsid Phosphoprotein: N protein; envelope protein: E protein; membrane glycoprotein: M protein; Copyright 2025, Elsevier Inc.
利用非同义突变与同义突变的比值来量化病毒的进化程度(承受的选择压力),发现北京的三波疫情期间SARS-CoV-2的Spike、N、ORF8、RdRp等蛋白编码区呈现显著的正选择压力,说明群体免疫压力驱动了病毒的适应性进化。
高风险突变增强病毒适应性

Fig. 5. Distribution of amino acid mutations in high-risk spike protein regions of SARS-CoV-2 detected by wastewater genomic surveillance. Copyright 2025, Elsevier Inc.
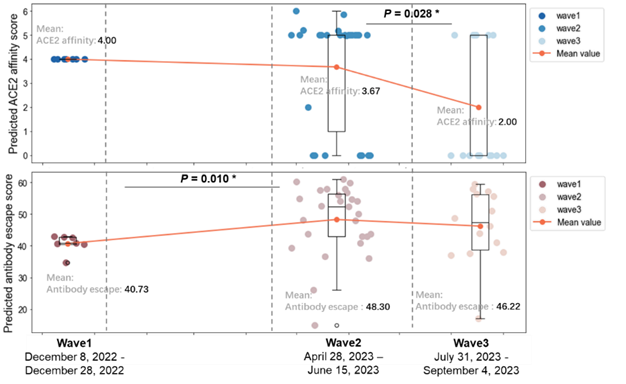
Fig. 6. VarEPS assessment of the strain risk index (a. Predicted ACE2 affinity score; b. Predicted antibody escape score) caused by spike protein amino acid mutations using wastewater monitoring data. Copyright 2025, Elsevier Inc.
进一步将不同位点的突变与其功能对应,发现多个Spike蛋白位点(如Ser371Phe、Leu452Arg)突变增强了病毒的ACE2结合能力,而Phe486Pro、Gln52His等位点突变显著提高了病毒的抗体中和逃逸潜能。整合所有的突变位点功能变化信息,基于VarEPS数据库分析,将病毒ACE2结合能力与抗体中和逃逸能力的变化水平量化表征,发现三波疫情期间优势变异株的中和逃逸能力显著增强,而ACE2亲和力则逐渐下降,说明抗体中和逃逸能力增强是北京新冠病毒的主要进化方向。
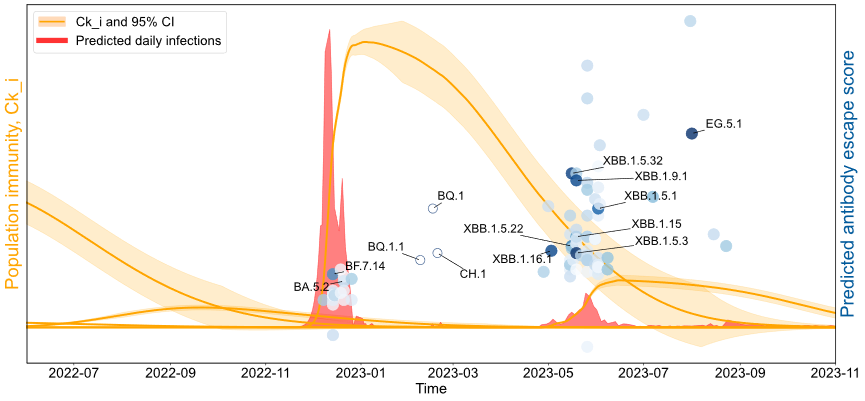
免疫轨迹与变异株替换关系

Fig. 7. Population immunity trajectories. The time-dependent population immunity (Cik) against variant i (BA.2.75, BA.5/BA.5.2, BF.7, BQ.1, BQ.1.1, XBB, XBB.1.5, CH.1.1, BN.1, XBB.1, EG.5.1, XBB.1.9, FL.1.*, BA.2.28, JN.1) induced by immune type k (CoronaVac ×3-inactivated vaccine, CoronaVac ×3-mRNA vaccine, CoronaVac ×4, BF.7 and BA.5.2 Infection, XBB lineage Infection). Trajectories for each immune class start at the dashed line. Copyright 2025, Elsevier Inc.
进一步将临床和污水监测结果及流调结果整合重新回溯北京疫情的发展规律,发现2022年12月疫情防控政策调整之前,由疫苗接种形成的群体免疫水平已大幅下降,使得政策调整之后BF.7.14毒株快速传播,引发第一波疫情并感染主城区超九成人口。大规模感染后短期内建立了对多种毒株(如BQ.1、CH.1.1等)的高水平群体免疫屏障,形成约3个月的“免疫空窗期”。但随着对XBB系列等新变异株的群体免疫水平衰减和其抗体中和免疫能力的提升,XBB系列毒株在2023年5月引发第二波疫情。图7清晰揭示了群体免疫水平与抗体中和逃逸毒株演替之间的时间关联关系,可为区域病毒演替规律解析提供重要依据。
小结
本研究基于污水病毒基因组监测、SNP变异谱分析与人群免疫衰减建模,系统揭示了集中感染背景下区域病毒加速进化的规律,为今后病毒预警系统的构建和疫苗更新策略提供了重要依据。